1. Covid-19 : Accès aux données
A l’heure des "fake news", ces fausses informations qui se répandent rapidement, il est important de vérifier en permanence l’exactitude des dépêches que nous recevons depuis divers supports, ce qui est loin d’être simple !
Les chercheurs de l’université Johns Hopkins ont dans ce but produit une carte qui permet de suivre et de visualiser les rapports sur l’épidémie en utilisant les données des Centres américains et chinois de contrôle et de prévention des maladies, de l’Organisation mondiale de la santé et d’autres sources. Au fur et à mesure de l’arrivée des rapports, la carte est actualisée avec le nombre total de cas confirmés, ainsi que ceux des décès et des guérisons. Chaque point rouge représente un foyer d’apparition de la maladie, dont la taille correspond à l’ampleur relative de contagion. En cliquant sur l’un d’entre eux, on obtient les informations relatives à la région.
Accès au site de l’université Johns Hopkins : https://systems.jhu.edu/
Accès aux données brutes sur leur espace github : https://github.com/CSSEGISandData/COVID-19
2. Objectif
Notre objectif est de développer une application Python pour :
-
récupérer les données brutes de la propagation du virus au format csv,
-
les filtrer par pays,
-
Tracer un graphic de l’évolution de la propagation
3. Moyens
-
Utilisez votre environement de développement préféré (idle, pycharm, spyder, … ) ou travailler en ligne avec https://repli.it
-
Clonez le dépôt de code à compléter sur classeroom.github : https://classroom.github.com/a/rzdhvt7c (A ne faire qu’une seule fois !)
4. Téléchargement des données
4.1. Téléchargement manuel
En fouillant dans le dépôt github de l’université Johns Hopkins, vous trouverez les données brutes des cas de contamination, de décès et de guérison :
Comme vous pouvez le constater, les données sont présentées sous forme de fichier csv, facilitant ainsi leur traitement par un tableur comme Microsoft excel ou LibreOffice Calc.
Nous reviendrons sur se format plus loin.
Pour le moment, concentrons nous sur le téléchargement d’un des fichiers. Je vous propose de télécharger le fichier time_series_19-covid-Confirmed.csv qui ressence les cas de contamination au quotidien depuis que les données épidémiologiques sont disponibles.
-
Pour télécharger le fichier, cliquez sur le lien du fichier, puis cliquez droit sur le bouton
Rawet choisirEnregistrer la cible du lien sous …. -
Ouvrez le fichier avec le tableur de votre choix. Il faut lui indiquer quel est le séparateur de champs, ici, c’est le caractère ",".
-
On peut maintenant utiliser notre tableur pour créer des graphique par exemple avec Excel :
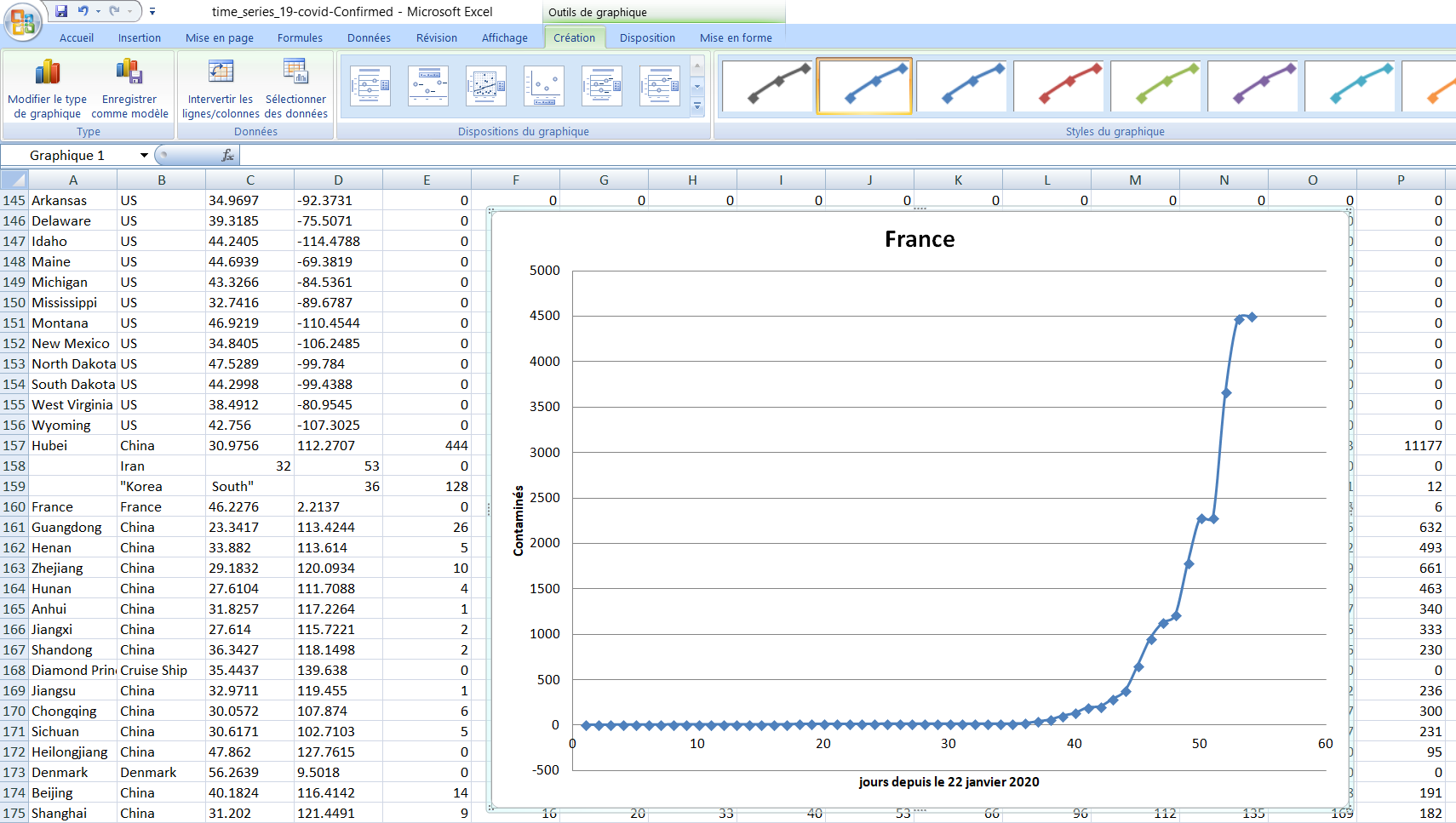
|
Il est important ici de remarquer les noms des colonnes qui constitue le tableau. On y trouve :
|
4.2. Fonction de téléchargement
Pour accéder à un fichier en ligne, nous aurons besoin du module urllib.request :
-
Ouvrez le fichier
covid_19v1.pyqui contient le code à compléter. -
Complétez le code ci-dessous en vous aidant des indications contenues dans les commentaires. Faites un usage approprié de l’aide fournie en lien dans le code.
-
Exécutez le et constatez le téléchargement du fichier dans le même dossier que votre script.
############################################
#
# Download a file
# Author : MS
# Date : 16/03/2020
#
# Replace the "___" by appropriate code
#
###########################################
import urllib.request as urllib2
def download_file(url, file_name):
'''
Download a file from url and copy it localy with file_name as name
'''
try:
# open url passed in argument
file = urllib2.urlopen(___)
# open file for writing in binary mode
# https://docs.python.org/3/library/functions.html#open
with open(___, '___') as output:
# write in output the file read
output.(file.___())
# OK : return true
return ___
except:
# Something is wrong : return false
return ___
# here the main code
if __name__ == '__main__':
# File name of datas
file = "time_series_19-covid-Confirmed.csv"
# where datas are located
url = ___
if download_file(url,file):
print(f'Téléchargement du fichier {file} terminé avec succès')
else:
print(f'Téléchargement du fichier {file} impossible')5. Lire le fichier csv
Le but de cette partie est de procéder à une lecture des données contenues dans le fichier CSV, notamment la listes des pays pour lesquels on en dispose.
|
Un fichier CSV (Comma-Separated Values) est un fichier tableur, contenant des données sur chaque ligne séparés par un caractère de séparation (généralement une virgule, un point-virgule ou une tabulation). Le format CSV est un format de texte simple qui est utilisé dans de nombreux contextes lorsque de grandes quantités de données doivent être fusionnées sans être directement connectées les unes aux autres. L’extension de ce type de fichiers est .csv, et ils peuvent être utilisés entre différents outils informatiques et bases de données. Des tableurs tels qu’Excel (Microsoft) ou Calc (LibreOffice) sont capables d’importer et exporter des fichiers CSV. Toutefois, en raison de sa structure basique, le format de fichier CSV ne convient que pour des données structurées simples. |
Pour accéder manipuler un fichier CSV, nous aurons besoin du module csv :
-
Ouvrez le fichier
covid_19v2.pyqui contient le code à compléter. -
Complétez le code ci-dessous en vous aidant des indications contenues dans les commentaires. Faites un usage approprié de l’aide fournie en lien dans le code.
-
Exécutez le et constatez l’affichage de la liste des pays et régions pour lesquels on dispose de données épidémiologiques.
############################################
#
# Read a csv file
# Author : MS
# Date : 16/03/2020
#
# Replace the "___" by appropriate code
#
###########################################
import urllib.request as urllib2
import csv
def download_file(url, file_name):
'''
Download a file from url and copy it localy with file_name as name
'''
try:
# open url passed in argument
file = urllib2.urlopen(___)
# open file for writing in binary mode
# https://docs.python.org/3/library/functions.html#open
with open(___, '___') as output:
# write in output the file read
output.(file.___())
# OK : return true
return ___
except:
# Something is wrong : return false
return ___
def read_CSV(file):
'''
Read a csv file and return :
- fields name as string in a list
- datas dictionary by countries in a list
'''
# datas is an empty list
datas = []
# open the file passed in argument as csvfile
# https://docs.python.org/3/library/csv.html#csv.reader
with open(file) as csvfile:
# Create a dictionnary with all datas
reader = csv.DictReader(____)
# each row in reader is a dictionary by country
for row in reader:
# add row in datas list
datas.___(___)
# return the list of dictionaries by countries
return datas
# here the main code
if __name__ == '__main__':
# File name of datas
file = "time_series_19-covid-Confirmed.csv"
# where datas are located
url = "https://github.com/CSSEGISandData/COVID-19/raw/master/csse_covid_19_data/csse_covid_19_time_series/time_series_19-covid-Confirmed.csv"
if download_file(url,file):
print(f'Téléchargement du fichier {file} terminé avec succès')
coutries = read_CSV(file)
print("liste des pays et régions recencés")
print("----------------------------------")
for row in coutries:
print(row['Province/State'], row['Country/Region'])
else:
print(f'Téléchargement du fichier {file} impossible')6. Filtrage par pays
On souhaite ici écrire une fonction qui recherche un pays ou une région pour ne fournir que ses propres données.
-
Ouvrez le fichier
covid_19v3.pyqui contient le code à compléter. -
Complétez le code de la fonction ci-dessous en vous aidant des indications contenues dans les commentaires.
-
Exécutez le en filtrant pour la France :
-
Province : France
-
Pays: France
-
-
Constatez l’affichage des données sous la forme d’un dictionnaire uniquement pour la France.
-
Exécutez le de nouveau en filtrant pour l'Italie puis pour la Chine, province de Hubei (https://fr.wikipedia.org/wiki/Hubei)
def data_for_country(data, state = "", country = ""):
# for each country in data
for pays in ___:
# if 'Province\State' key match with state given in argument
# and if 'Country/Region' key match with country given in argument
if pays[___] == ___ and pays[___] == ___:
return pays
# here the main code
if __name__ == '__main__':
# File name of datas
file = "time_series_19-covid-Confirmed.csv"
# where datas are located
url = "https://github.com/CSSEGISandData/COVID-19/raw/master/csse_covid_19_data/csse_covid_19_time_series/time_series_19-covid-Confirmed.csv"
if download_file(url,file):
print(f'Téléchargement du fichier {file} terminé avec succès')
countries = read_CSV(file)
print("liste des pays et régions recencés")
print("----------------------------------")
# for row in coutries:
# print(row['Province/State'], row['Country/Region'])
france = data_for_country(countries, 'France', 'France')
print(france)
else:
print(f'Téléchargement du fichier {file} impossible')7. Tracer le graphe de la propagation
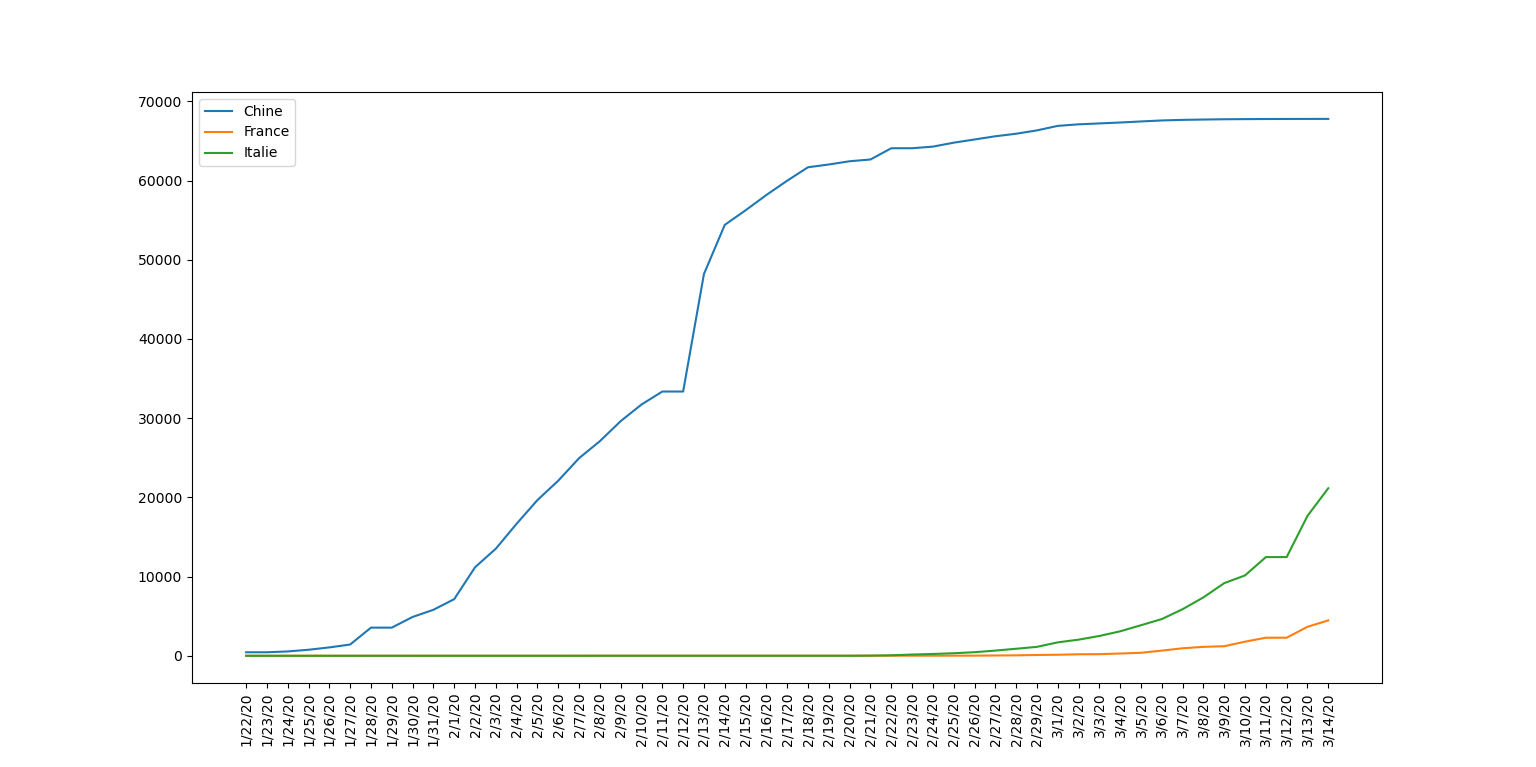
On souhaite maintenant tracer l’évolution du nombre de personnes dont l’infection est confirmée pour la Chine (Hubei), la France et l’Italie afin de visualiser et comparer la cinétique de la propagation dans ces trois pays.
Nous allons pour cela utiliser le module matplotlib.
|
Le module Documentation du module : https://matplotlib.org/ Un très bon tutoriel |
-
Ouvrez le fichier
covid_19v4.pyqui contient le code à compléter. -
Complétez le code de la fonction ci-dessous en vous aidant des indications contenues dans les commentaires, de la documentation du module et/ou du tutoriel.
-
Exécutez le en filtrant pour la Chine, province de Hubei, l'Italie et la France:
-
Observez l’affichage des courbes de la propagation des cas confirmés d’infection.
-
Ajoutez de nouveau pays comme par exemple, les Etats Unis, l’Angleterre, l’Espagne, ….
-
Tracer les graphiques de l’évolution du nombre de décès pour les pays de votre choix.
-
Tracer les graphiques de l’évolution du nombre de guéris pour les pays de votre choix.
-
Ajouter un titre à vos graphique (comment se dit "titre" en anglais ?).
def trace_data_for_country(country):
'''
Prepare lists of datas for x and y axis
'''
# x is an empty list
x = ___
# y is an empty list
y = ___
# browse through the country dictionary to get datas.
# - Keys contain datas for x axis (dates)
# - Values contain datas for y axis (number of daily cases)
#for key, value in country.___():
for key, value in country.items():
# Filter inappropriate keys
if key != 'Province/State' and key !='___' and key != '___' and key != '___':
# Add key to the x list
x.___(___)
# add value to the y list.
# value must be an integer
y.___(int(___))
# return a tuple of lists x,y
return x,y
# here the main code
if __name__ == '__main__':
# File name of datas
file = "time_series_19-covid-Confirmed.csv"
# where datas are located
url = "https://github.com/CSSEGISandData/COVID-19/raw/master/csse_covid_19_data/csse_covid_19_time_series/time_series_19-covid-Confirmed.csv"
if download_file(url,file):
print(f'Téléchargement du fichier {file} terminé avec succès')
countries = read_CSV(file)
print("liste des pays et régions recencés")
print("----------------------------------")
# for row in coutries:
# print(row['Province/State'], row['Country/Region'])
chine = data_for_country(countries, 'Hubei', 'China')
france = data_for_country(countries, 'France', 'France')
italie = data_for_country(countries, '', 'Italy')
print(chine)
print(france)
print(italie)
# Figure dimensions
plt.figure(figsize=(10, 7))
# Rotate ticks and labels of the x-axis
plt.xticks(rotation=90)
# Plot datas for China
x , y = trace_data_for_country(chine)
plt.plot(x, y, label="Chine")
# Plot datas for France
___
___
# Plot datas for Italy
___
___
# Add title to graph : "Infectés"
___
# Add legend to graph
plt.___()
# Show graph
plt.___()
# Save the figure as '19-covid-Confimed.png'
plt.___('9-covid-Confimed.png')
# Close graph
plt.___()
else:
print(f'Téléchargement du fichier {file} impossible')8. Résutats attendus
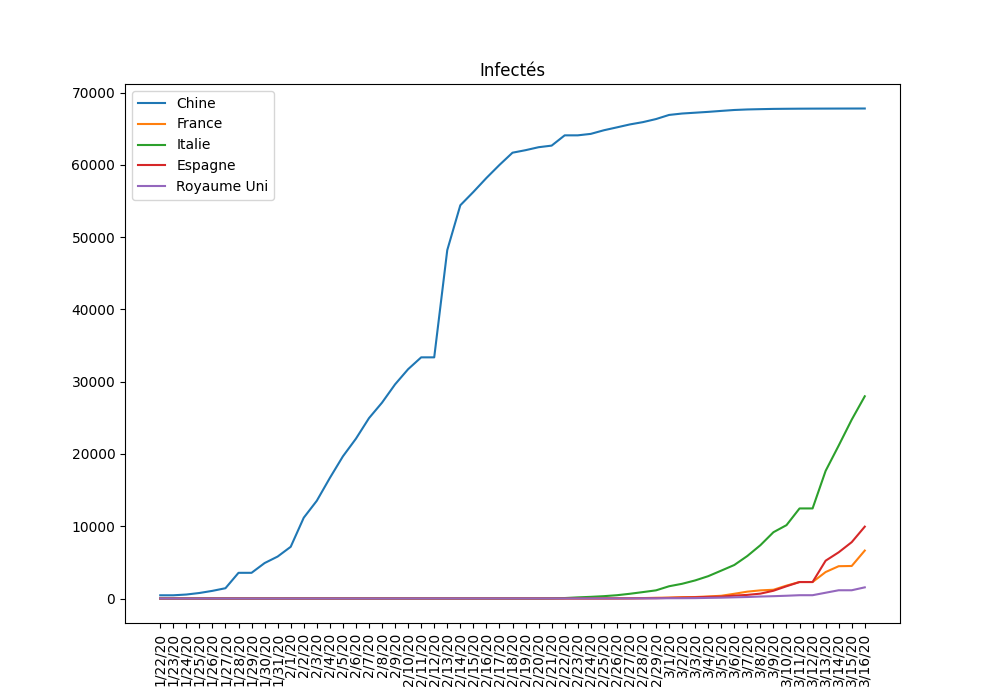
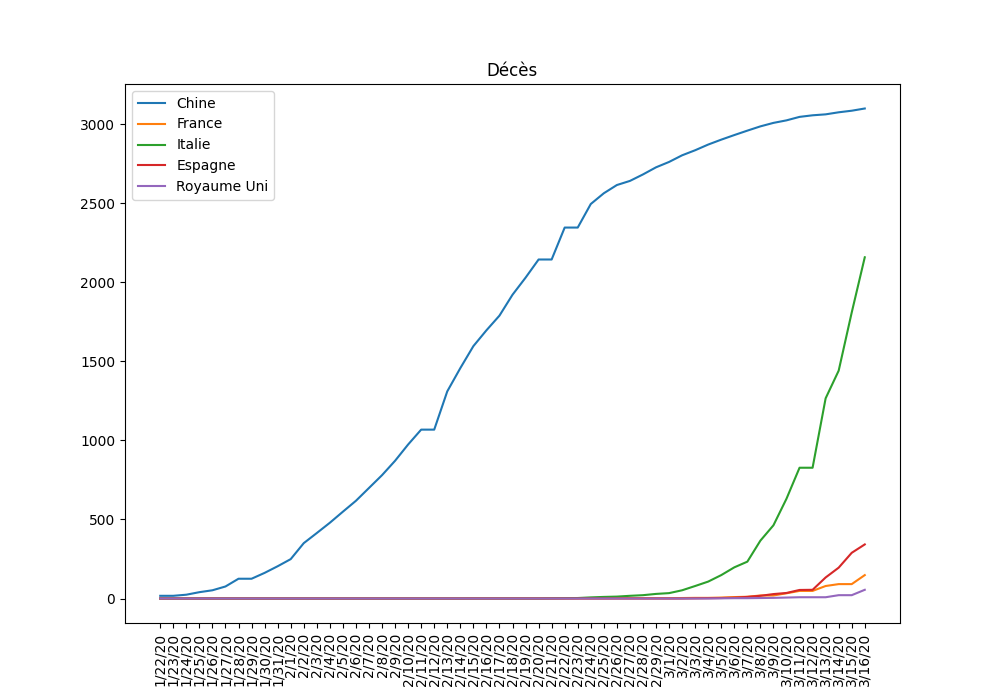
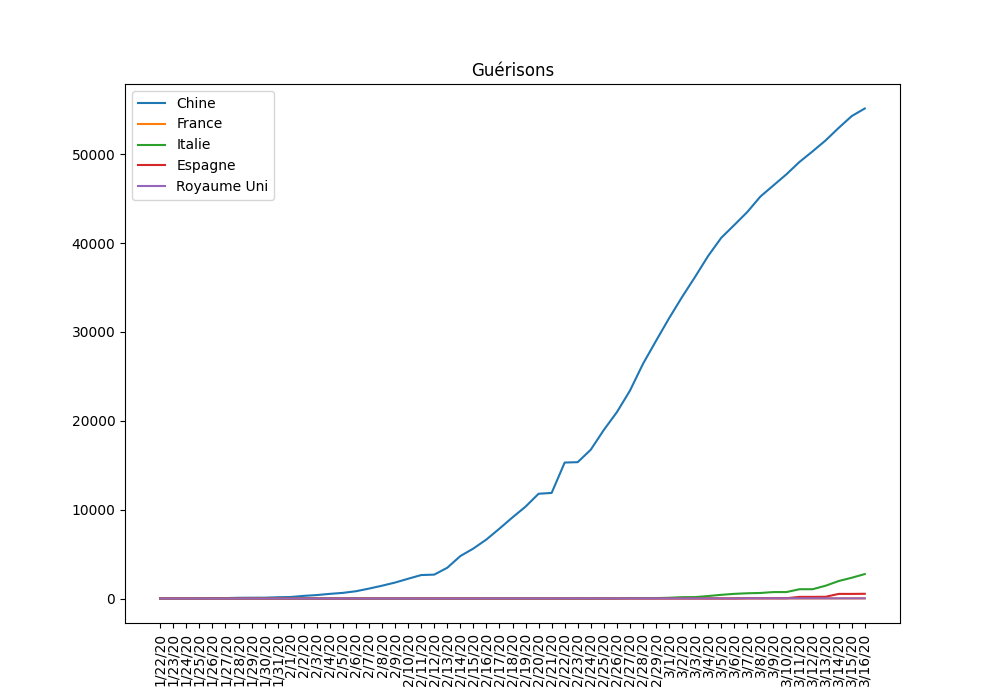
9. Conclusion
Les graphiques permettent de comprendre la cinétique de la propagation du virus au sein de la population. Les situations dans divers pays sont comparables avec quelques jours de décalages.
La comparaison avec Hubei, la province de chine la plus formtement touchée est intéressante. En effet, sa population est de 59 millions d’habitants ce qui est comparable à la population française et italienne, cependant, sa supperficie est de 186 000 km², soit presque 4 fois plus petit que la France et 1,6 fois plus petit que l'Italie.
On remarque que plus la densité de population est importante, plus la propagation est rapide.
Il est donc important de suivre les recommendations d’éloignement social pour limiter la propagation du virus et donc le pic de contamination.
En Chine, il semblerait que le confinement total que l’État a mis en place a réussi à contrôler l’épidémie. Ces mesures drastiques ne sont pas encore utilisées en France, mais l’éloignement social peut permettre d’aplatir la courbe plus rapidement, pour que toute la population ne tombe pas malade en même temps et que le système de santé puisse prendre en charge les malades sans être débordé.
